PCTI 230. Clonación del gen del patógeno Vibrio parahaemolyticus para su posible producción como vacuna en plantas
Carlos Angulo
Autor de Correspondencia
Dr. Héctor Nolasco Soria
Editor
16/02/2024
Fecha de Aprobación
Biotecnología y Ciencias Agropecuarias
Categoría
Autores
Hassian León, Carlos Angulo.
Centro de Investigaciones Biológicas del Noroeste, S.C. (CIBNOR), Grupo de Inmunología y Vacunología. eangulo@cibnor.mx
La vibriosis en peces, crustáceos y moluscos, o la gastroenteritis en humanos, es una problemática de salud causada principalmente por el patógeno Vibrio parahaemolyticus
Abstract
The appearance of multidrug-resistant strains of Vibrio parahaemolyticus in seafood has given rise to the generation of recombinant vaccines through genetic engineering as an alternative. The objective was to generate a genetic construct that expressed a protein to produce a vaccine against V. parahaemolyticus. The geneVp was ligated to the pBi121 vector, generating a pBi121::genVp construct that was cloned into Escherichia coli and subsequently into Agrobacterium tumefaciens. Positive clones were confirmed by endpoint PCR with a 1,100 bp amplicon. In conclusion, the genetic construct pBi121:genVp was correctly generated and transferred to A. tumefaciens. Therefore, its use in plants is proposed to produce the protein and evaluate its immunogenicity in an animal model.
Keywords: vaccine design, enteric pathogen, recombinant protein
Resumen
La aparición de cepas multidrogoresistentes de Vibrio parahaemolyticus en alimentos marinos ha dado como alternativa la generación de vacunas recombinantes mediante ingeniería genética. El objetivo fue generar una construcción genética que expresara una proteína para producir una vacuna contra V. parahaemolyticus. El genVp se ligó al plásmido pBi121, generando una construcción pBi121::genVp que se clonó en Escherichia coli y posteriormente en Agrobacterium tumefaciens. La clonas positivas se confirmaron mediante PCR de punto final con un amplicón de 1,100 pb. En conclusión, la construcción genética pBi121: genVp fue generada y transferida correctamente a A. tumefaciens. Por lo que se propone su uso en plantas para producir la proteína y evaluar su inmunogenicidad en un modelo animal.
Palabras clave: diseño de vacunas, patógeno entérico, proteína recombinante.
Problemática
Usuarios
Proyecto
Introducción
Los peces y mariscos son consumidos en el mundo. (Parlapani, 2021). Sin embargo, estos alimentos pueden contaminarse con patógenos, entre los que destaca Vibrio parahaemolyticus (Letchumanan et al., 2019). Esta bacteria produce toxinas hemolisina directa termostable (TDH), hemolisina relacionada con TDH (TRH) y la proteína TLH que provocan vómito, diarrea aguda y hasta la muerte (Hong et al., 2018). Estudios recientes demuestran que existen alternativas contra V. parahaemolyticus, entre ellas la vacunación (Mdarhri et al., 2022; Ji et al., 2020). Las vacunas recombinantes son una alternativa actual para el tratamiento de las enfermedades transmitidas por alimentos (ETA). Estas vacunas se producen mediante ingeniería genética, que consiste en una construcción elaborada con ADN circular (plásmido) y un gen que expresa una proteína con la capacidad de estimular el sistema inmune (Daniali et al., 2024). En su producción comúnmente se utiliza el plásmido pBi121, que emplea la maquinaria genética de células vegetales para la producción de la vacuna recombinante (Monreal-Escalante et al., 2019).
Objetivos
Generar la construcción genética (pBi121::genVp) que codifique para un antígeno compuesto de los factores de virulencia de V. parahaemolyticus para su posible expresión en plantas.
Materiales y métodos
Dos cultivos independientes de Escherichia coli TOP 10, que contenían las construcciones pUC57::gen_VP y el plásmido pBi121, respectivamente, se propagaron en medio LB con kanamicina 50 mg/L durante 2 días a 37 oC. Posteriormente, la extracción de ADN plasmídico se realizó por el método de lisis alcalina (Sambrook et al, 1989). A continuación, la digestión enzimática del plásmido pUC57 se realizó a 37 oC durante 20 minutos con SmaI-SacI para abrir el plásmido y liberar el “gen_VP” (Monreal-Escalante et al., 2019). Los productos de la digestión fueron separados en una electroforesis en gel de agarosa al 1% a 75 V por 40 minutos, donde las bandas cortadas se purificaron conforme al kit QIAEX II Gel Extraction (QIAGEN®) y los productos se resguardaron a -20 oC. La reacción de ligación con la enzima T4 ADN ligasa se llevó a cabo durante 24 horas a 4 oC, uniendo el gen_VP con el plásmido pBi121 para generar la construcción pBi121::gen_VP (Villegas, 2023).
Para replicar la construcción genética pBi121::gen_VP, células de E.coli TOP10 (Invitrogen® C404003TM) se transformaron mediante el método de choque térmico, sembrando las células en medio LB con kanamicina a 50 mg/L e incubándolas a 37 oC durante 2 días. Posteriormente, se extrajo la construcción genética por un método alcalino, seguido de una PCR de punto final para confirmar la presencia del gen_VP (Sambrook et al, 1989). A continuación, la transformación de Agrobacterium tumefaciens GV-3101 con la construcción pBi121::gen_VP se realizó por electroporación a 1.8 V, sembrando las células en medio YM con kanamicina a 50 mg/L e incubándolas a 28 oC durante 4 días. Finalmente, se realizó otra extracción alcalina de la construcción genética y posteriormente una PCR de punto final para confirmar las clonas positivas (Monreal-Escalante et al., 2019) (Figura 1).
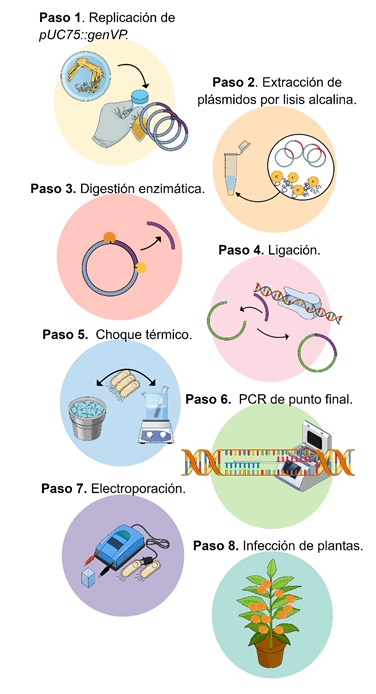
Figura 1. Los 8 pasos generar una construcción genética. Paso 1: replicación de pUC57::gen_Vp. Paso 2: extracción por lisis alcalina. Paso 3: liberar el gen_Vp y abrir el plásmido pBi121 con enzimas. Paso 4: ligar el gen_Vp con el plásmido pBi121. Paso 5: transferir pBi121::gen_Vp a E.coli por choque térmico. Paso 6: confirmar la transferencia por PCR punto final. Paso 7: transferir pBi121::gen_Vp a A. tumefaciens mediante electroporación. Paso 8: infectar plantas A. tumefaciens con pBi121::gen_Vp.
Resultados y Discusión
Con la liberación del gen_VP del plásmido comercial pUC57 (Figura. 2) y su inserción en el plásmido pBi121 se generó la construcción pBi121::gen_VP. La construcción genética se clonó en E.coli para replicarla y así obtener suficientes copias; para posteriormente clonarla en A. tumefaciens; y finalmente confirmar las clonas positivas mediante PCR de punto final, obteniendo como resultado en ambos casos un amplicón de 1,100 pb (Figura. 3). La construcción genética pBi121::gen_VP se elaboró con el propósito posterior de utilizarla para la transformación de plantas, ya que las células vegetales son utilizadas como plataforma para la expresión de proteínas recombinantes en la producción de vacunas orales (Monreal-Escalante et al., 2019).
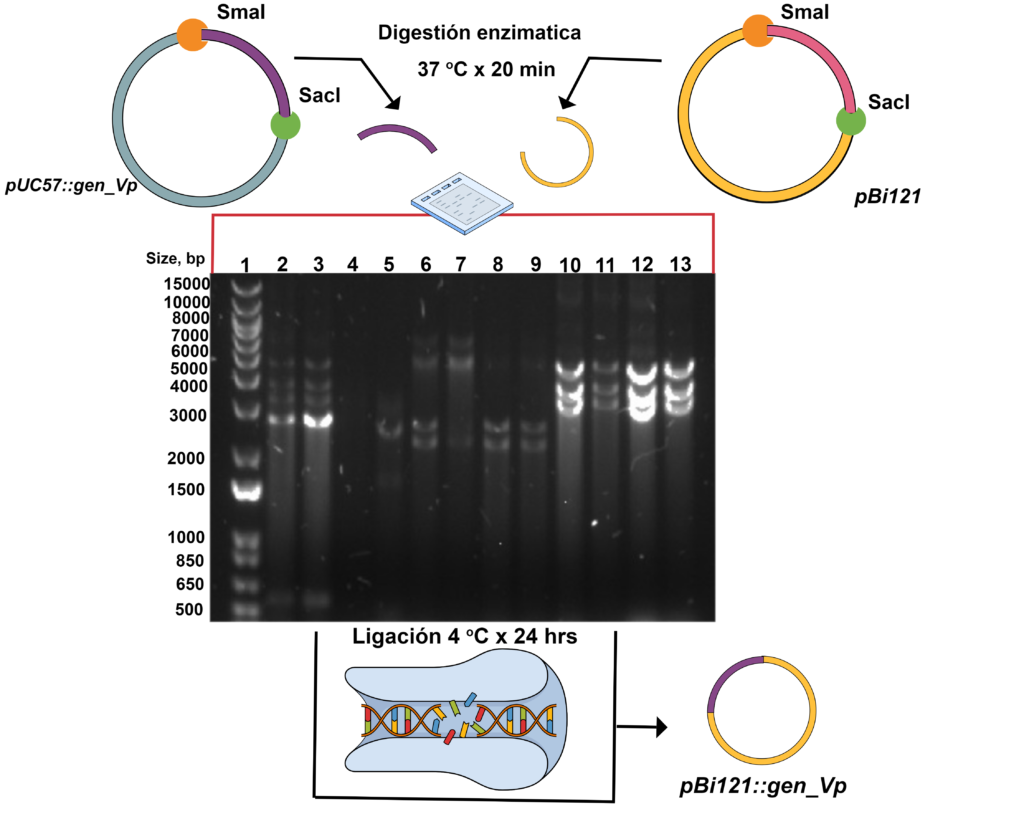
Figura 2. Perfil de restricción del pUC57::gen_VP y de pBi121. Carril 1: marcador 1 kb plus. Carriles 3-6: liberación del gen_Vp de 2,300 pb. Carriles 7-10: apertura del plásmido pBi121con un tamaño de 12,000 pb.
Conclusión
La construcción genética pBi121::gen_VP se generó con base en un gen de un antígeno compuesto de los factores de virulencia de V. parahaemolyticus, que fue posible clonar en E. coli y en A. tumefaciens para su uso en la transformación genética de plantas para la expresión de la vacuna recombinante.
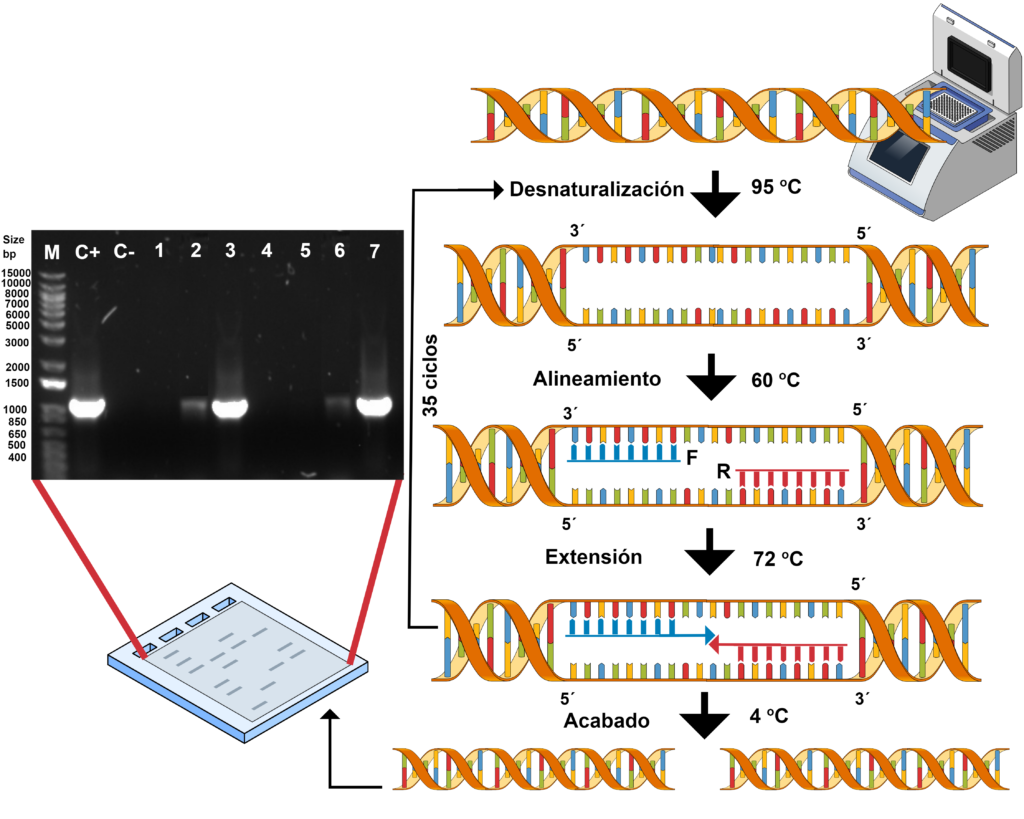
Figura 3. Producto de PCR de clonas de E. coli TOP10. Carril M: marcador de peso molecular 1 kb plus. Carril C+: control positivo. Carril C-: control negativo. Carriles 2, 3, 6 y 7: amplicon 1,100 pb del gen_VP.
Impacto Socioeconómico
Impacto socioeconómico
La generación de vacunas contra la vibriosis en peces, crustáceos y moluscos, o la gastroenteritis en humanos, es una alternativa para combatir los daños que causa el patógeno Vibrio parahaemolyticus en la producción de alimentos para el consumo humano. Además, la producción de vacunas recombinantes en plantas con el propósito de su administración oral probablemente tendría un impacto mayor porque se ha justificado por su bajo costo, fácil distribución, sencilla administración y potencial estabilidad funcional a temperatura ambiente.
Referencias
Parlapani, F. (2021) Diversidad microbiana de los mariscos. Opinión actual en ciencia de los alimentos. 37, 45-51. https://doi.org/10.1016/j.cofs.2020.09.005
Letchumanan, V., Ab, S., Hei, S., Chan, K., & Lee, H. (2019) Determination of antibiotic resistance patterns of Vibrio parahaemolyticus from shrimp and shellfish in Selangor, Malaysia.. Progress in Microbes and Molecular Biology,2(1), 1–8. DOI: http://10.36877/pmmb.a0000019.
Hong, T., Yanagawa, H., Thuan, K., Hara-Kudo, Y., Taniguichi, T., & Hayashidani, H. (2018) Prevalence of Vibrio parahaemolyticus in seafood and water environment in the Mekong Delta, Vietnam. The Journal of Veterinary Medical Science. 80(11);1737-1742. doi: 10.1292/jvms.18-0241.
Mdarhri, H., Benmessaoud, R., Yacoubi, H., Seffar, L.,Guennouni, H., Hamam, M., Boussettine, R., Filali-Ansari, N., Lahlou, F., Diawara, I., Ennaji, M., & Kattani-Halabi, M. (2022) Alternatives Therapeutic Approaches to Conventional Antibiotics: Advantages, Limitations and Potential Application in Medicine. Antibiotics(Basel).16;11(12):1826. doi: 10.3390/antibiotics11121826.
Ji, Q., Wang, S., Ma, J., & Liu, Q. (2020) A review: progress in the development of fish Vibrio spp. Vaccines. Immuno Lett. 226; 46-54. doi:10.1016/j.imlet.2020.07.002.
Daniali, M., Mousavi, T., & Abdollahi, M. (2024) Biological products in medicine. Encyclopedia of Toxicology. 2;4, 117-132. https://doi.org/10.1016/B978-0-12-824315-2.00039-7
Monreal-Escalante, E., Rosales-Mendoza, S., Govea-Alonso, D., Campa-Córdova, Á., & Angulo, C. (2019) Genetically-engineered plants yield an orally immunogenic PirA-like toxin from Vibrio parahaemolyticus. Int J Biology Macromolecules. 137; 126-131 doi:10.1016/j.ijbiomac.2019.06.159
Sambrook, J., Fritsh, E., & Maniatis, T. (1989) Molecular Cloning: Laboratory Manual (2nd ed). Cold Spring Harbor Laboratory Press.
Villegas, A. (2023) “Diseño y desarrollo de una vacuna recombinante multiepitópica basada en las toxinas TDH, TRH Y TLH de Vibrio parahaemolyticus para su expresión en plantas y microalgas. Universidad Autónoma de México. Tesis de Licenciatura.
