PCTI 220. Plataforma tecnológica de análisis genético multiespecie de última generación para potenciar el desarrollo acuícola de Baja California Sur, México
Ricardo Pérez Enríquez
Autor de Correspondencia
Dr. Héctor Nolasco Soria
Editor
08/02/2023
Fecha de Aprobación
Biotecnología y Ciencias Agropecuarias
Categoría
Autores
Ricardo Pérez Enríquez1, Cristina Escobedo-Fregoso2, Gabriela Mendoza Carrión1, Adriana Max3, Susana Ávila1
1Centro de Investigaciones Biológicas del Noroeste, S.C. (CIBNOR), 2CONACYT-CIBNOR, 3Posgrado-CIBNOR.
Abstract
Genetic management programs in aquaculture species require the monitoring of the genetic diversity and inbreeding levels for their optimization. In the present work, we developed a technological platform known as “2bRAD” that would be simultaneously applicable to the economically important species of Baja California Sur. The results indicated the technical feasibility of the genetic characterization of marine fishes (snapper, almaco jack, totoaba) and oyster with a low-density panel of genetic markers. For shrimp, the panel was of medium size with a potential use in other type of genetic analyses. The economic feasibility of its application by larvae and juvenile producing companies is being evaluated.
Keywords: genetic selection, DNA, molecular markers, aquaculture.
Resumen
Los programas de manejo genético de las especies acuícolas requieren del monitoreo de diversidad genética y endogamia para optimizar su productividad. En este trabajo se desarrolló una plataforma tecnológica de análisis llamada “2bRAD” que pueda ser aplicable simultáneamente en las especies acuícolas de interés comercial de Baja California Sur. Los resultados indicaron la factibilidad de la técnica para realizar la caracterización genética con un panel de marcadores de baja densidad para peces marinos (pargo, jurel, totoaba) y ostión. Para el camarón se obtuvo un panel de mediana densidad con potencial para otro tipo de análisis genéticos. La factibilidad económica para su aplicación por parte de las empresas productoras de larvas y juveniles, está siendo evaluada.
Palabras clave: selección genética, ADN, marcadores moleculares, acuacultura.
Problemática
Usuarios
Proyecto
Introducción
El sector acuícola es uno de los de mayor crecimiento en Baja California Sur. Se estima que, en 2019, el cultivo de especies como el camarón, el ostión y peces marinos (por ejemplo, pargo, jurel, totoaba), entre otras, representó un valor de casi $500 millones de pesos empleando a más de 1,350 personas en el estado (CSABCS, comunicación personal). Con el fin de potenciar esta producción, las empresas acuícolas requieren implementar programas de manejo y selección genética basados, entre otros elementos, del monitoreo de la diversidad genética, la endogamia y del pedigrí, ya que un manejo sin controles adecuados puede llevar a un bajo desempeño en la producción (Gjedrem 2010). Una de las herramientas tecnológicas necesarias para ello, es la que utiliza el ADN para la obtención de la huella genética de los organismos de cultivo. Esta huella, basada en marcadores tipo “SNPs” (polimorfismos de nucleótidos simples), es utilizada para caracterizar la composición y diversidad genética, los niveles de endogamia, el parentesco dentro de lotes de producción y la diferenciación entre líneas de cultivo (Maqsood y Ahmad 2017) (Fig. 1).
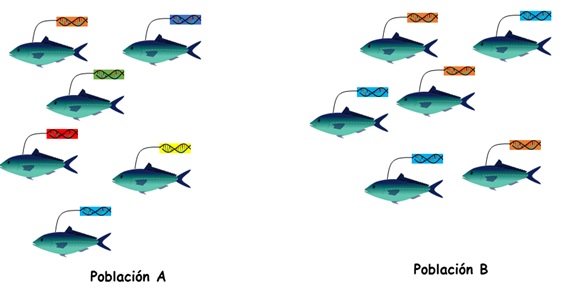
Fig. 1. Líneas de cultivo con distintos niveles de diversidad genética.
Dependiendo de estos indicadores, las unidades de producción de larvas y semillas estarán en condiciones de tomar mejores decisiones de reproducción y manejo de sus lotes de cultivo. En México, hay estudios que han probado la utilidad de los SNPs para la caracterización genética de poblaciones de camarón de cultivo (Perez-Enriquez et al. 2018), silvestres (Mejía-Ruiz et al. 2021) y análisis de parentesco (Perez-Enriquez y Max-Aguilar, 2016). Sin embargo, dado que no se cuenta con las herramientas de aplicación rutinaria en nuestro país, se depende de enviar las muestras al extranjero para el procesamiento de laboratorio.
Objetivo
Desarrollar una plataforma de análisis basada en paneles de marcadores genéticos de baja densidad (100 a 300 SNPs), de aplicación simultánea para especies de crustáceos, moluscos y peces de importancia acuícola para el Estado de Baja California Sur.
Materiales y métodos
La plataforma de análisis multiespecie se basa en la técnica llamada 2bRAD (Wang et al. 2012) que consiste en la obtención de SNPs después de cortar el ADN con “tijeras moleculares” (enzimas de restricción propiamente dichas) y unir a los fragmentos resultantes en secuencias sintéticas de ADN que contienen códigos de barra de identificación individual, lo que permite mezclar el ADN de muchos organismos durante la secuenciación masiva (MiSeq, Illumina) (Fig. 2).
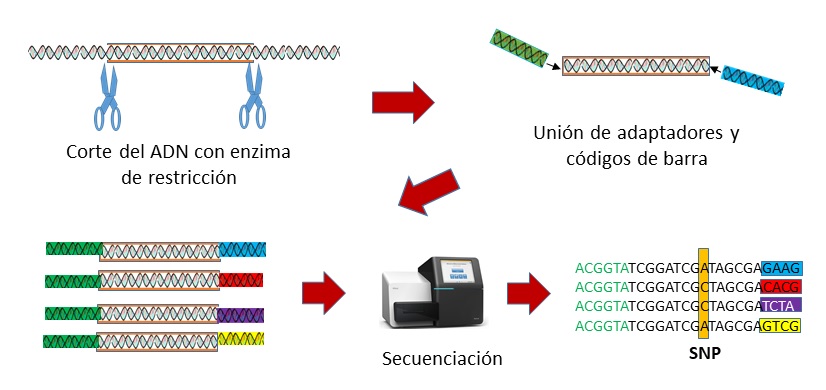
Fig. 2. Diagrama simplificado de la técnica 2bRAD para descubrimiento de SNPs.
La caracterización genética posterior se realiza mediante análisis bioinformáticos utilizando como referencia los genomas disponibles en el banco mundial de datos genómicos “GenBank” del Instituto Nacional de Salud de los EE.UU (https://www.ncbi.nlm.nih.gov), así como programas especializados como GenAlEx (Peakall y Smouse, 2012), Cervus (Kalinowski et al., 2007) y Colony (Jones y Wang, 2010). El estudio se dividió en tres etapas: 1) análisis bioinformático para la selección de la enzima de restricción a utilizar; 2) secuenciación para la evaluación de la técnica en muestras representativas; 3) secuenciación para la validación de la plataforma con muestras procedentes de cultivos. Los análisis se llevaron a cabo en los laboratorios de Genética Acuícola y de Genómica y Bioinformática del CIBNOR. Las especies seleccionadas en la primera y segunda etapas fueron el camarón blanco Penaeus (Litopenaeus) vannamei, el jurel Seriola rivoliana y el ostión del Pacífico Crassostrea gigas. En la tercera etapa se incluyeron, además de las anteriores y a manera de análisis exploratorio, el huachinango Lutjanus peru, el pargo lunarejo Lutjanus guttatus y la totoaba Totoaba macdonaldi.
Resultados y Discusión
El análisis bioinformático con 4 enzimas (BsaXI, BcgI, AlfI y CspCI), indicó que la enzima BcgI presentó la mayor homogeneidad en el número de sitios de reconocimiento en el genoma de las especies y que, utilizando dos adaptadores con dos bases selectivas cada uno, sería posible obtener un panel de baja densidad con un número adecuado de marcadores genéticos. 2) La secuenciación de los genomas digeridos de las especies de interés con la enzima BcgI confirmó que el número de marcadores tipo SNPs que pueden obtenerse, utilizando dos adaptadores con dos bases selectivas cada uno, ronda entre 300 y 400 SNPs para el jurel y el ostión; siendo potencialmente útiles para análisis de diversidad genética y parentesco. Para el camarón, el número sobrepasa los 2,800 SNPs, que si bien abre la posibilidad a otro tipo de análisis, no sería factible como plataforma de baja densidad; por ello, esta especie requiere de una aproximación metodológica distinta. 3) El análisis de la validación indicó que el número de SNPs obtenido fue adecuado para la caracterización genética de las especies de estudio y, particularmente, para los análisis de parentesco de jurel y ostión ( 3). Los análisis exploratorios con el huachinango, el pargo lunarejo y la totoaba indicaron el potencial de la misma tecnología para la caracterización genética de lotes de cultivo.
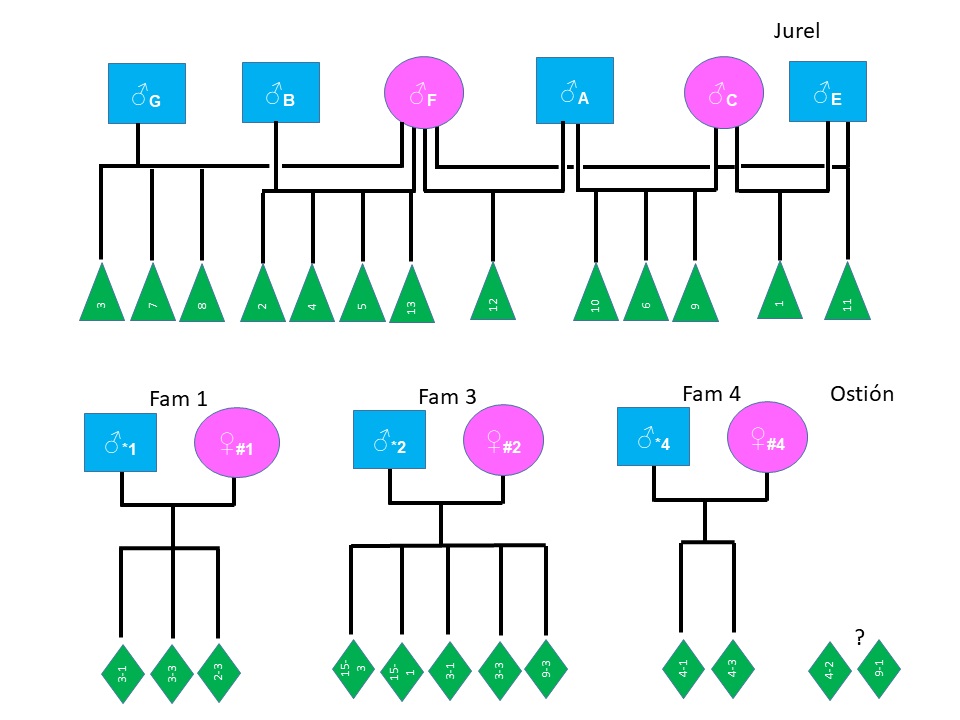
Fig. 3. Análisis de parentesco basados en perfiles genéticos 2bRAD para jurel (arriba) y ostión (abajo). Los triángulos y rombos corresponden a las progenies. Los ejemplares marcados con signo de interrogación no fueron asignados a ninguna familia particular.
Conclusiones
Los resultados indican que la plataforma 2bRAD es útil para la caracterización genética de líneas de cultivo de diversas especies de importancia comercial (principalmente ostión y peces marinos), no sólo de Baja California Sur, sino también para otras regiones del país. Para el camarón, el grupo de trabajo del CIBNOR está desarrollando una aproximación tecnológica distinta.
Impacto Socioeconómico
Las empresas acuícolas que requieran optimizar sus programas de entrecruzamiento y selección tendrán disponibilidad de utilizar esta herramienta tecnológica a nivel nacional, no teniendo que enviar sus muestras al extranjero. Para ello, la factibilidad económica para su aplicación se está analizando.
Referencias
Gjedrem, T. (2010) Selection and Breeding Programs in Aquaculture. Springer. Dordrecht, The Netherlands. 364 pp.
Jones OR, Wang J (2010) COLONY: a program for parentage and sibship inference from multilocus genotype data. Molecular Ecology Resources, 10: 551–555.
Kalinowski ST, Taper ML, Marshall TC (2007) Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 16: 1099–1106.
Maqsood HM, Ahmad SM (2017) Advances in Molecular Markers and Their Applications in Aquaculture and Fisheries. Genetics of Aquatic Organisms, 1: 27–41
Mejía-Ruíz P, Perez-Enriquez R, Mares-Mayagoitia JA, Valenzuela-Quiñonez F (2020) Population genomics reveals a mismatch between management and biological units in green abalone (Haliotis fulgens). PeerJ, 8:e9722.
Peakall R, Smouse PE (2012) GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research – an update. Bioinformatics, 28: 2537–2539.
Perez-Enriquez R, Max-Aguilar A (2016) Pedigree traceability in whiteleg shrimp (Litopenaeus vannamei) using genetic markers: A comparison between microsatellites and SNPs. Ciencias Marinas, 42: 227–235.
Perez-Enriquez R, Robledo D, Houston RD, Llera-Herrera R (2018) SNP markers for the genetic characterization of Mexican shrimp broodstocks. Genomics, 110: 423–429.
Wang S, Meyer E, McKay J K, Matz MV (2012) 2b-RAD: A simple and flexible method for genome-wide genotyping. Nature Methods, 9: 808–810.
