PCTI 147. DIVERSIDAD GENÉTICA DE LA CABRA SANTANDEREANA (CAPRA HIRCUS) MEDIANTE MICROSATÉLITES: MODELO APLICABLE PARA LA CONSERVACIÓN Y APROVECHAMIENTO GENÉTICO DE LAS CABRAS NATIVAS MEXICANAS
Lady Katerine Serrano Mujica
Autor de Correspondencia
Dr. Héctor Nolasco Soria
Editor
13/12/2014
Fecha de Aprobación
Biotecnología y Ciencias Agropecuarias
Categoría
Autores
Lady Katerine Serrano Mujica1, Cesar Ascanio Hernández1, Javier Enrique Vargas-Bayona2, Ángela Jiménez Leaño2, Víctor Arcila Quiceno2, Cesar Serrano Novoa2, Julia Teresa Bedoya2.
Universidad Cooperativa de Colombia 1Facultad de Medicina Veterinaria y Zootecnia, sede Bucaramanga, 2Grupo de Investigación en Ciencias Animales.
Lady.Serranom@campusucc.edu.co
En centro América y sur América, la producción de cabras se concentra en regiones áridas donde prevalecen la pobreza, la escasez de agua y las sequías.
Abstract
The native races were originated in America by natural selection, as a result of genetic isolation subjected for centuries, after having been introduced by the conquerors. In the department of Santander,Colombia, there is a goat (Capra hircus), a naturalized animal genetic resource, whose population is estimated to be 5,000 animals. The aim of this study was to establish a first approach to understanding the genetic resource, using microsatellite molecular markers, which allows to recognize and take advantage of the races own biological characteristics, making this study a model for implementation in Mexican native goats in order to take advantage of the genetic heritage for years managed to be adapted to the different geographic regions of Mexico.
Keywords: molecular biology, conservation, native, animal genetic resource.
Resumen
Las razas nativas en América se originaron por selección natural como consecuencia del aislamiento genético al que fueron sometidas durante siglos, después de haber sido introducidas por los conquistadores. En el departamento de Santander, Colombia, existe la cabra santandereana (Capra hircus) un recurso zoogenético naturalizado, cuya población se estima que es de 5000 ejemplares. El objetivo de este estudio fue establecer una primera aproximación al conocimiento del recurso genético, mediante la utilización de marcadores moleculares tipo microsatélite, que permita reconocer y aprovechar características biológicas propias de las razas, convirtiendo este estudio en un modelo a seguir para implementar en las cabras nativas mexicanas, con el objetivo de aprovechar el patrimonio genético que durante años lograron adaptarse los diferentes regiones geográficas del territorio mexicano.
Palabras clave: biología molecular, conservación, nativas, recurso zoogenético
Problemática
Usuarios
Proyecto
Introducción
En el mundo, más de 2000 millones de personas habitan zonas de condiciones climáticas adversas, muchas de ellas se dedican a la producción de especies animales rústicas como la cabra (LADA/FAO 2003). En centro América y sur América, la producción de cabras se concentra en regiones áridas donde prevalecen la pobreza, la escasez de agua y las sequías durante gran parte del año Echavarría et al. (2006).
Objetivos
El objetivo del proyecto fue, mediante el empleo de marcadores moleculares tipo microsatélite, determinar la diversidad genética en y entre núcleos de cabras santandereanas (Capra hircus) (Fig. 1, Vargas 2007), y comparar dicha diversidad genética con razas foráneas, para establecer una primera aproximación al conocimiento genético, bajo la hipótesis de existencia de correlación entre la variación genética y la viabilidad de la población. Por ello, la necesidad de cuantificar dicha diversidad (variabilidad), para así poder realizar una mejor y más acertada política de conservación. La metodología implementada en este estudio podrá ser aplicada para reconocer la diversidad (variabilidad) de las cabras nativas mexicanas, al igual que identificar el origen genético y si existe alguna asociación entre grupos genéticos que dieron origen a la cabra santandereana, teniéndose como base que los países fueron colonizados por españoles los cuales llegaron a tierras Americanas con diversidad de especies tanto animales y se esparcieron libremente por todo el nuevo continente. Los estudios de diversidad dan como resultado una aproximación a las características biológicas tales como: la adaptación, aclimatación y resistencia a parásitos, entre otras variables y bondades que hacen importante la conservación del patrimonio genético nativo. En la conservación de los recursos genéticos, el objetivo principal consiste en preservar la variabilidad dentro de las poblaciones, bajo la hipótesis de existencia de correlación entre la variación genética y la viabilidad de la población. Por ello, la necesidad de cuantificar dicha diversidad (variabilidad), para así poder realizar una mejor y más acertada política de conservación.

Figura 1. Cabra Santandereana (Vargas 2007).
Materiales y Métodos
Se colectaron 85 muestras de 8 ml de sangre (tomada de la vena yugular de las cabras) en tubos con anticoagulante EDTA (7.2 mg de K3 por cada 4 ml de sangre) y almacenadas a una temperatura entre 4ºC a 6ºC. De estas muestras, 65 fueron tomadas de cabras santandereanas, seleccionadas, de acuerdo a las características fenotípicas propias de la raza, y las otras 20 muestras fueron tomadas de razas foráneas (Boer, Alpina americana, Alpina francesa, Toggenburg y Sannen). El procesamiento de las muestras se realizó en el laboratorio de Biología Molecular de la Universidad Cooperativa de Colombia sede Bucaramanga. Se extrajo el ADN cromosomal, empleando el kit DNA 2000® (Corpogen SA) siguiendo las instrucciones del fabricante, y se cuantificaron las muestras por fluorometría (QubitFluorometro®). Se realizaron reacciones de PCR multiplex (agua, DNTPs, buffer 10x, MgCl2, iniciador forward, iniciador reverse, Taq polimerasa y ADN molde) siguiendo la metodología descrita por Acosta y col. (2005) y Barrera y col. (2006), amplificando 13 microsatélites (CNS1S, BM1824, ILSTS005, BM1818, INRA005, ETH10, ETH25, HAUT27, INRA063, ITLS030, MAF65, OARFCB304, CSRD247) previamente reportados, en estudios de diversidad genética en cabras criollas (Acosta et al 2005, Chacón et al 2010, Serrano et al 2009), altamente informativos. Los 1105 amplificados fueron visualizados en geles de agarosa (Sigma®) al 1.5%, para determinar si las temperaturas de anillamiento de la PCR eran las óptimas. Además, fueron evaluados por electroforesis (47°C, 1263 voltios, 52 Miliamperios, durante 2 horas) en geles de poliacrilamida (12% PAA) Sigma®, en condiciones desnaturalizantes (con urea a una concentración de 2%). Se utilizaron marcadores de peso molecular de 100pb (CorpoGen). Los geles fueron teñidos con nitrato de plata (Sigma®) y revelados con hidróxido de sodio (Sigma®) para la visualización de los alelos Ariza (2012), en los cuales se diferenciaron alelos homocigotos y heterocigotos de acuerdo a Serrano y Ascanio (2013) (Fig. 2).
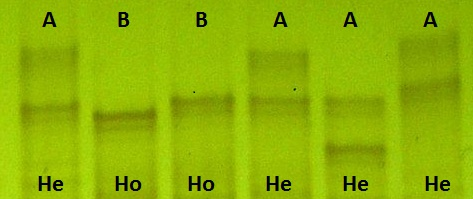
Figura 2. Los carriles de electroforesis pertenecen A: cabra santandereana B: alpina francesa, visualizándose alelos Ho: homocigotos (una sola banda intensa) y He: heterocigotos (dos bandas intensas) en geles de poliacrilamida bajo condiciones desnaturalizantes, teñidos con nitrato de plata (Serrano y Ascanio 2013).
Con base en la movilidad electroforética de cada alelo, se calculó el número de pares de bases de cada uno, utilizado como referencia los marcadores de peso molecular. Los alelos se genetipificaron en una base de datos en Microsoft Excel®, que sirvió para el análisis con el software bio-informatico Genetix® (Mendez et al 2013) que permitió obtener algunos índices de variabilidad como el Índice de contenido polimórfico (PIC), Heterocigosidad Observada (Ho) y Heterocigosidad esperada (He).
Como parte de los resultados se encontró que 12 de los microsatélites presentaron alto grado de polimorfismo, ya que se encontraron de 6 a 8 alelos diferentes por microsatélite, lo que indica una elevada diversidad alélica, coincidiendo con el número mínimo de alelos reportado por Barker et al (1994). El microsatélite ETH10 fue igual para todas las muestras presentando solo dos alelos, siendo poco informativo en estudios de diversidad en cabras nativas (Estévez et al 2003); en contraste, Rodríguez (2010) reportó a este microsatélite como altamente polimórfico. Se toma en cuenta que un número mínimo de tres alelos por microsatélite reduce errores en el cálculo de distancia genética (Barker et al 1994). El número medio de alelos encontrado en este estudio fue de 5.5, similar a lo reportado por Carrera (2005) para cabras nativas brasileras, superior a lo reportado por Azor y col. (2008) en cabras criollas Chilenas (5.1) y por Muema y col. (2009) en cabras de África Subsahariana (3.82) evaluadas en Ujiji y Guinea Bissau, e inferior a lo encontrado por Acosta y col. (2005) quienes reportaron 6.38 alelos en cabra Mejorera. Los estudios de diversidad con microsatélite son importantes ya que permiten hacer un análisis filogenético y la búsqueda de genes útiles, entre otras variables como el grado de parentesco entre los individuos de una misma especie. La cabra santandereana resultó diferente entre individuos ubicados en la misma región geográfica, lo que indica no tener un grado de consanguineidad tan marcado, lo que no se esperaba ya que el cruzamiento entre individuos en su mayoría es por selección natural. La cabra santandereana resultó no ser tan próxima a las razas foráneas estudiadas lo que es un indicador positivo, ya que afirma que la influencia de estas razas no ha afectado la diversidad. Por otra parte, el número de alelos heterocigotos (Fig. 2) determinaron la heterogosidad media observada que fue del 53.5%, y el 78% de los marcadores estudiados mostraron valores superiores al 50%, que fue indicador de una elevada variabilidad genética. De los 13 microsatélite empleados en este estudio, 11 microsatélite (CNS1S, BM1824, ILSTS005, BM1818, INRA005, INRA063, ITLS030, MAF65, OARFCB304, CSRD247, HAUT27) resultaron muy informativos con valores de PIC superiores a 0.5. Entre la heterogosidad media y el PIC existe una relación, ya que cuando uno sufre un aumento el otro también lo hace (Vaiman et al 1994), lo que indica que el muestreo fue adecuado y confirma la calidad de los marcadores. Diez (CNS1S, BM1824, ILSTS005, BM1818, INRA005, HAUT27, INRA063, MAF65, OARFCB304, CSRD247) de los trece microsatélites demostraron estar en equilibrio, y esto acontece cuando los alelos del locus están distribuidos al azar en la población y no existe asociación entre el par de alelos que un hijo recibe de sus padres; según la prueba de Hardy Weinberg, dice que en una población grande bajo apareamiento aleatorio, sin selección, mutación o migración, las frecuencias alélicas y genotípicas permanecen constantes de generación en generación. Los locus desequilibrados están presentes en poblaciones domesticas ya que estas están sometidas a selección artificial siendo una de las principales razones de desequilibrio (Azor et al 2008). El número de pares de bases (pb) encontrado por el análisis electroforético, para cada microsatelite se muestra en la Tabla 1.
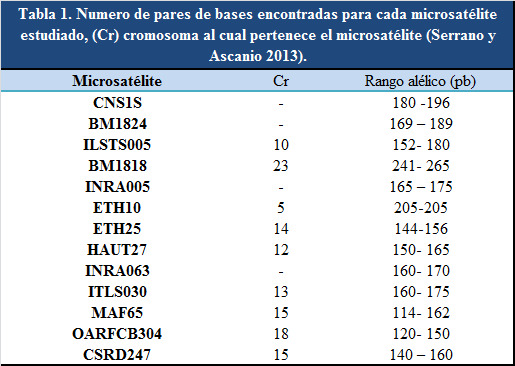
La diferencia entre alelos varía en el número de repeticiones. Los resultados obtenidos concuerdan con lo reportado por Chacón y col. (2010), y difiere a lo reportado por Acosta y col. (2005) quienes describen al microsatélite BM1824 como monomórfico con 169pb y al ETH10 como polimórfico con 205- 215pb, comportándose diferente a lo encontrado en este estudio, lo que puede indicar mayor diversidad de alelos.
Los resultados más importantes fueron que la población nativa presentó 166 alelos, comparada con las razas foráneas estudiadas que solo presentaron 34 alelos, lo que indica un alto grado de diversidad, haciendo de gran interés esta raza para ser conservada; no obstante, para mantener los recursos nativos durante generaciones se deben desarrollar programas de apareamiento evitando la consanguineidad, que es el cruzamiento entre individuos emparentados, que se traducen genéticamente en un incremento de individuos (homocigotos) como consecuencia una depresión consanguínea, es decir, una reducción de los valores medios fenotípicos de los caracteres productivos y reproductivos, y por último, y como consecuencia de esos problemas reproductivos, la disminución inevitable o la extinción. A pesar de que existe la técnica moderna de electroforesis capilar con fluorometría para los estudios de diversidad (Chacón et al 2010). La más utilizada es la electroforesis en geles de poliacrilamida (Ariza 2012) bajo condiciones desnaturalizantes, dando óptimos resultados a un bajo costo. Los marcadores moleculares de tipo microsatélite, son una excelente elección debido a que ciertas características que son dominantes, tienen alto número de alelos en el mismo locus, son fáciles de analizar, y son abundantes en el genoma de los eucariotas, lo que permite identificar poblaciones con una diversidad genética reducida (generalmente más vulnerables a posibles cambios ambientales), e identificar subpoblaciones genéticamente diferenciadas. Al ser una técnica relativamente fácil y sensible, sería aplicable para estudiar la diversidad de las razas de cabras nativas mexicanas, con el fin de conservar estos recursos genéticos.
La metodología propuesta para el estudio de la diversidad genética de las cabras nativas mexicanas consiste en la colecta de sangre de la población nativa, con un número mínimo de 65 animales muestreados de la raza nativa y 20 muestras provenientes de razas foráneas, realizando cada uno de los pasos indicados en este estudio, hasta la obtención de ADN cromosomal. Se aplica la batería de microsatélites reportados en este estudio, interpretado los datos que van a determinar el grado de diversidad de las cabras nativas y la influencia de las cabras foráneas sobre estas razas, permitiendo determinar el patrimonio genético.
Impacto Socioeconómico
La identificación de núcleos puros de las cabras nativas mexicanas, permitirá conocer su origen filogenético y el aprovechamiento de características biológicas propias significativamente importantes como la rusticidad (adaptación) para adopción de mejores sistemas de producción, que ayudarán al crecimiento económico en el país, incentivando a la conservación y preservación de los recursos genéticos nativos de México.
Referencias
Acosta JM., Martínez A., Pestaño A., Brown RP., Delgado JV. (2005). Caracterización genética de la cabra majorera de Fuerteventura con Microsatélites. Arch. Zootec. 54: 261-266.
Ariza F. (2012). Entrevista, docente de la Universidad Nacional de Colombia sede Bogotá, 9-16 de mayo de 2012.
Azor, P. J., Valera, M., Sarria, J., Avilés, J. P., Nahed, T. J., Delgado, M. P. & Castel, J. M. (2008). Estimación de las relaciones genéticas entre razas caprinas españolas y criollas utilizando microsatélites. Revista ITEA. 2: 323.
Barker F. (1994). A global protocol for determining genetic distances among domestic livestock breeds. World Congress on Genetic Applied to Livestock Production. 21: 501-508.
Barrera, G. P.; Martínez R.; Pérez J. E.; Polanco N.; Ariza F. (2006). Evaluación de la variabilidad genética en Ganado Criollo Colombiano mediante 12 marcadores microsatélite. AGRI 38: 35-45.
Carrera, M. M. P. (2005). Variabilidade e relações genéticas entreraças caprinas nativas brasileiras, Ibéricas e canárias. Universidades Federale da Paraíba, Pernanbuco y Ceará. Tesis Dr. Cs.16 – 24.
Chacón, E., Martínez A.; Vásquez. F.; Perez E.; Delgado J. (2010). Caracterización genética de la cabra criolla cubana mediante microsatélite. Revista cubana de Ciencias Agrícolas, vol 44: 221-226.
Echavarría, F.; Gutiérrez, R.; Ledesma, R.; Banuelos, R.; Aguilera, J.; Serna, P. (2006). Influence of small ruminant grazing systems in a semiarid range in the State of Zacatecas Mexico: I Native vegetation. Técnica Pecuaria en México 44: 203-217.
Estévez, J. y Montero, M. (2003). Análisis de los restos animals del Edificio Alfa del Puig Morter de Son Ferragut (Sineu, Mallorca). In: Castro, P.Y., Escoriza, T. y Sanahuja, M” E. Mujeres y hombres en espacios domésticos. Trabajo y vida social en la Prehistoria de Mallorca (c. 700-500 cal ANE). El Edificio Alfa del Puig Morter de Son Ferragut (Sineu, Mallorca): 336-361.
LADA/FAO. (2003). Agenda prospectiva de investigación y desarrollo tecnológico para la cadena productiva cárnica ovino-caprina en Colombia. Ministerio de agricultura y desarrollo rural. Proyecto de transición de la Agricultura. TECNOS. Fundación Andina para el Desarrollo Tecnológico y Social. Bogotá, D.C.115-116.
Méndez J., Portilla P., Rincón X., Dickson L., D Aubeterre R. (2013). Diversidad genética en la cabra criolla Venezolana mediante análisis con microsatélite. Revista Científica, FCV-LUZ / Vol. XXIII, 3: 238-244.
Muema, E. K., Wakhungu, J. W., Hanotte, O. & Jianlin, Han. (2009). Genetic diversity and relationship of indigenous goats of Sub-saharan Africa using microsatellite DNA markers.Livestock Research for Rural Development. 2: 2.
Rodríguez. Jorge. (2004). Determinación de parentesco por medio de análisis de ADN microsatélite en alpacas (lama pacos). Tesis para optar título de Médico Veterinario. Lima (Perú). Universidad Nacional Mayor de San Marcos.10: 7.
Serrano. L.K., Ascanio.C.A. (2013). Diversidad genética de la cabra santandereana (capra hircus) mediante el empleo de marcadores moleculares tipo microsatélite. Teses para optar el título de Médico Veterinario y Zootecnista. Bucaramanga (Colombia). Universidad Cooperativa de Colombia.12: 50.
Serrano M, Calvo J H, Martínez M, Marcos-Carcavilla A, Cuevas J, González C, Jurado J J and Díez de Tejada P. (2009). Microsatellite based genetic diversity and population structure of the endangered Spanish Guadarrama goat breed. BMC Genetics, Volume10, 10ñ:1186/1471-2156-10-61
Vaiman, D., Mercier, D., Moazami-Goudarzi, K., Eggen, A.,Ciampolini, R., Lepingle, A., Velmala, R., Kaukinen, J., Varvio,S. L. & Martin, P.(1994). A set of 99 cattle microsatellites: characterization, synteny mapping, and polymorphism. Mammalian Genome 5.pag.288.
