PCTI 119. ANÁLISIS DE INFORMACIÓN GENÓMICA: INVESTIGACIÓN BIOINFORMÁTICA (CIBNOR)
Eduardo Romero Vivas
Autor de Correspondencia
Dr. Héctor Nolasco Soria
Editor
30/12/2012
Fecha de Aprobación
Ingenierías
Categoría
Autores
Eduardo Romero Vivas, Fernando D. Von Borstel Luna, Joaquín Gutiérrez Jagüey y Ricardo Vázquez Juárez
Centro de Investigaciones Biológicas del Noroeste, S.C.
Las tecnologías de secuenciación de DNA generan un gran volumen de datos que involucra el almacenamiento, manejo e interpretación, lo que se constituye en uno de los grandes retos en este campo.
Abstract
Great volumes of data generated by current research on genomics and proteomics possess particular storage, manage and processing challenges, which bioinformatics address. Within this context, in this paper the demands imposed by these challenges are considered, for the design of bioinformatics facilities at CIBNOR, and the creation of a bioinformatics research group. Knowledge of important commercial aquaculture species genomes, and its potential application, has been benefited by the integration of bioinformatics into current research at CIBNOR.
Keywords: bioinformatics, node, genomic information.
Resumen:
La investigación actual en las ciencias genómicas y proteómicas conlleva a la generación de un gran volumen de datos y consecuentemente genera grandes retos para su almacenamiento, manejo e interpretación; siendo la bioinformática la disciplina propuesta para resolver estos retos. En este contexto, este artículo describe la estrategia de integración y consolidación de un grupo de investigación, y el diseño de un nodo computacional de bioinformática que permitan satisfacer las demandas en esta área. La integración de la Bioinformática con los actuales proyectos en el CIBNOR ha potenciado la aplicación del conocimiento del genoma de importantes especies acuícolas de interés comercial y de gran impacto socio-económico.
Palabras clave: bioinformática, nodo, información genómica.
Problemática
Usuarios
Proyecto
Introducción
Para obtener el primer borrador de la secuencia de DNA del genoma humano, constituido con aproximadamente 3 mil millones de pares de bases, se requirieron 13 años de trabajo y una inversión de 3 mil millones de dólares; éste fue publicado en 2003. La figura 1 muestra dicha cantidad de información en volúmenes impresos. Actualmente, las tecnologías de secuenciación de nueva generación permiten secuenciar esa cantidad en tan solo una semana, al punto que el genoma completo del Dr. James Watson (premio Nobel y descubridor de la doble cadena del DNA, junto con el Dr. Francis Crick), se pudo obtener en dos meses en el año 2008, con una redundancia de 7.4 veces y un costo menor al millón de dólares (Nature, 2009).

Figura .1 Muestra una fotografía tomada en el museo de la medicina de la fundación Wellcome, en ella se aprecia un librero que contiene la secuencia completa del genoma humano, en 120 volúmenes representando los 24 cromosomas.
El uso de las tecnologías de secuenciación de nueva generación ha revolucionado la biología molecular impactando en áreas como la académica, médica, farmacéutica, biotecnológica, agroquímica y en la industria alimentaria. Es importante señalar que la evolución de la bioinformática es un proceso dinámico y que en pocos años se ha convertido en el punto de convergencia de diversos campos de la biología moderna; de tal manera que se requieren de especialistas que satisfagan las demandas en las áreas de programación, transcriptómica, bases de datos, aplicaciones, proteómica, sistemas y genómica, entre otras.
Objetivos
Con este antecedente, el objetivo fue la integración de un Grupo de Bioinformática conformado por especialistas en los campos de matemáticas, computación y biología, que desarrollará la capacidad de interacción entre los diferentes grupos especializados como la genómica, proteómica y transcriptómica (en las ciencias “-ómicas”), entre otras, para abordar los retos en el análisis de la información generada. Así mismo se propuso la creación del Nodo Computacional de Bioinformática (Fig. 2) en el Centro de Investigaciones Biológicas del Noroeste, S.C. (CIBNOR), que permita satisfacer las necesidades de cómputo intensivo y de alto rendimiento que se requieren para este tipo de investigaciones.
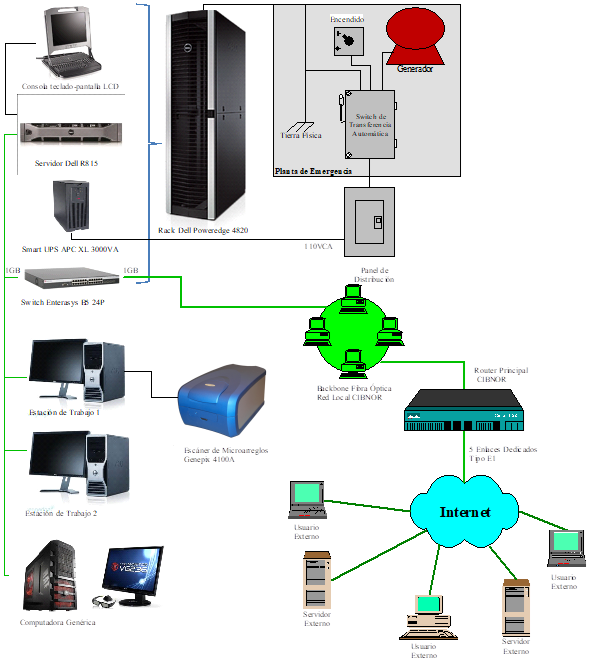
Figura 2. Esquema de conectividad del Nodo de Bioinformática en el CIBNOR. En la parte superior izquierda se muestra el equipamiento de cómputo y red desglosado fuera del gabinete principal. En la parte superior derecha el circuito de la planta eléctrica de emergencia. En la parte inferior izquierda se muestran las estaciones de trabajo y el escáner de microarreglos. En la parte inferior derecha la conectividad de los enlaces de red e internet.
Materiales y Métodos
El Grupo de Bioinformática se ha capacitado por medio de cursos y talleres nacionales e internacionales, y coordina los cursos de “Métodos computacionales en bioinformática” y “Cómputo científico” en el posgrado del CIBNOR. El grupo es responsable académico del Nodo Computacional de Bioinformática: lugar de convergencia de los análisis y repositorio de la información generada.
La selección de infraestructura software y hardware se proyectó en 3 etapas. En la primera se creó la infraestructura de soporte: un switch, una fuente de poder ininterrumpible, e instalaciones eléctricas y de red de cómputo (Fig. 3) con gran ancho de banda, indispensable para el traslado de grandes bases de datos genómicas. En una segunda etapa, se montó un servidor multi-núcleo, con alta capacidad de memoria RAM y un arreglo de discos duros. Se adquirió un escáner de microarreglos para estudiar respuesta transcriptómica global y se complementó con 2 estaciones de trabajo con tarjetas GPU (Graphic Processor Unit) para el desarrollo de aplicaciones de procesamiento en paralelo, tal como el análisis de expresión genética en microarreglos (Romero-Vivas, et al. 2012).

Figura 3. Vistas parciales del nodo computacional de bioinformática, donde se aprecia la infraestructura de soporte del lado izquierdo y del lado derecho una toma más a detalle del servidor principal y su consola.
Dichas capacidades se obtuvieron gracias al proyecto de investigación “Aplicación de la genómica funcional como estrategia para la mejora continua de la industria del camarón” (SAGARPA-2009-C02-126427). En una tercera etapa se incrementará la capacidad para tareas de ensamblaje de novo con la expansión de memoria y adquisición de más servidores para procesamiento en paralelo. El nodo ofrece más de 500 programas específicos que permiten realizar pre-procesamiento de secuencias, ensamblaje, alineamiento de secuencias, anotación funcional, generación de árboles filogenéticos en bacterias y análisis de expresión génica, entre otros. El uso de estos programas se complementa con las librerías genómicas públicas de las cuales se cuenta con copias locales para su análisis y procesamiento en el nodo.
Conclusiones
En conclusión, a través de los trabajos experimentales en el CIBNOR, y el análisis de la información genómica generada en el Nodo de Bioinformática, se ha incrementado el conocimiento del genoma de importantes especies acuícolas de interés comercial y de gran impacto socio-económico, lo cual permitirá que se puedan lograr herramientas in-situ para la toma decisiones preventivas, disminuyendo posibles pérdidas en cultivos; y generar el conocimiento que permita mejorar las especies para una mayor producción.
Impacto Socioeconómico
Impacto Socioeconómico
Como consecuencia de la revolución tecnológica que se ha dado durante la última década en las ciencias biológicas, gracias a las herramientas de última generación de las que se obtiene un gran volumen de datos, el procesamiento de los datos crudos y su análisis representa una gran parte del esfuerzo experimental. Debido a la dramática disminución por los costos de secuenciación por el auge de las plataformas de secuenciación masiva, el costo por el análisis y procesamiento de información (bioinformática) representará muy pronto, la mayor proporción en relación al costo global en las investigaciones biológicas (Sboner, et al.2011). Por ejemplo, el cultivo de camarón (L. vannamei), ha tenido un incremento importante en los dos últimos años (2011 y 2012), pero esta actividad económica se ha visto seriamente afectada por el Virus de la Mancha Blanca que ha provocado una caída drástica de la producción. A pesar de esto, todavía no se cuenta con el genoma completo de esta especie de cultivo. En el CIBNOR, se están desarrollando microarreglos, como una herramienta genómica que eventualmente permitirá identificar oportunamente cuando el camarón de granja está siendo afectado por enfermedades como el Virus de la Mancha Blanca (Fig. 4).

Figura 4. Microarreglo para Camarón de granja diseñado en el CIBNOR. En la porción derecha se muestra un acercamiento. Cada punto representa un gen y el color señala su participación ante una condición específica: Amarillo – neutro, verde – sub expresado, rojo – sobre expresado.
De igual forma, el grupo de bioinformática participa en proyectos de investigación genómica que estudian los mecanismos de diferenciación sexual de la almeja “Mano de León”, orientados a aumentar su producción (Llera, et al.2010).
Referencias
Nature. 2009. Ed. Information overload, Nature 460, 551 (30 July 2009) | doi:10.1038/460551a; Published online 29 July 2009
Sboner A., Mu X. J., Greenbaum D., Auerbach R.K., y Gerstein M.B. 2011. The real cost of sequencing: higher than you think!, Genome Biology 2011, 12:125 http://genomebiology.com/2011/12/8/125
Romero-Vivas E., Von Borstel F. D., y Villa-Medína I. 2012. Implementación en GPU del Estadístico t para análisis de expresión genética en microarreglos. Acta Universitaria. Universidad de Guanajuato 2012, 22:6, pp. 23-30
http://www.actauniversitaria.ugto.mx/index.php/acta/article/view/370
Llera-Herrera Raul, Garcia-Gasca Alejandra, Romero-Vivas Eduardo, Arnaud Huvet, Ana M Ibarra. 2010. Identification and expression analysis of gametogenic-related genes in the ovary and testis of the hermaphrodite lion-paw scallop Nodipecten subnodosus. Physiomar 2010.
