PCTI 156. IDENTIFICACIÓN DE BIOMARCADORES PROTEÓMICOS DEL INTESTINO DEL MOSQUITO ANOPHELES ALBIMANUS TRANSMISOR DE LA MALARIA EN MÉXICO, INFECTADO CON PLASMODIUM BERGHEI.
Vania Verónica Serrano Pinto
Autor de Correspondencia
Dr. Héctor Nolasco Soria
Editor
14/12/2014
Fecha de Aprobación
Medicina y Ciencias de la Salud
Categoría
Autores
Vania Verónica Serrano Pinto
Centro de Investigaciones Biológicas del Noroeste, S.C.
vserrano04@cibnor.mx
El principal vector para la transmisión de la malaria en las costas de México, infectado por el protozoario parásito P. vivax, es el mosquito Anopheles albimanus.
Abstract
The main vector for transmission of malaria in the coasts of Mexico is the Anopheles albimanus mosquito. The midgut of disease-transmitting mosquitoes carries out a variety of functions that are related to blood feeding. The objective was to analyze the midgut of An. albimanus infected with Plasmodium berghei using a proteomic approach to identify proteins over expressed following Plasmodium invasion. The two major classes of proteins were overexpressed on the immunity/defense and digestive protease groups.
Key Words: Malaria, proteomics, midgut, Anopheles albimanus, Plasmodium berguei.
Resumen
El principal vector para la transmisión de la malaria en las costas de México es el mosquito Anopheles albimanus. El intestino de los mosquitos transmisores de enfermedades lleva a cabo una variedad de funciones que están relacionadas con la alimentación a base de sangre. El objetivo del estudio fue analizar el intestino de An. albimanus infectado con Plasmodium berghei utilizando análisis proteómicos para identificar proteínas que se sobre expresan después de la invasión con Plasmodium. Las dos principales clases de proteínas sobre expresadas fueron las de inmunidad y defensa y el grupo de proteasas digestivas.
Palabras clave: Malaria, proteómica, intestino, Anopheles albimanus, Plasmodium berguei.
Problemática
Usuarios
Proyecto
Introducción
El principal vector para la transmisión de la malaria en las costas de México, infectado por el protozoario parásito P. vivax, es el mosquito Anopheles albimanus. Las hembras del mosquito Anopheles son las únicas que se alimentan de sangre, elemento necesario para el proceso de vitelogénesis (Osta et al 2004, Xu et al 2006); por lo tanto, son también las responsables de la transmisión de la enfermedad. La sangre se digiere en el intestino de este mosquito y es el primer lugar de interacción con el parásito. En este sitio, mediante la activación de diversos mecanismos del sistema inmune innato, se bloquea casi por completo el desarrollo del parásito debido a la lisis en el epitelio del intestino y a la melanización temprana de los oquistes. No obstante lo anterior, algunos oquistes sobreviven en el lumen y en el epitelio del intestino del vector (Collins 1994), por lo que es indispensable eliminarlos. En mosquitos se tienen identificadas tres vías de transducción de señales relacionadas con inmunidad, las vías Toll, IMD y la vía JAK/STAT. La información relacionada con las proteínas responsables de la respuesta inmune en mosquitos es limitada (Dimopoulos et al 1998), debido a lo cual, la determinación de la función de estas proteínas podría contribuir al entendimiento de las respuestas selectivas de la inmunidad innata en este insecto, con la finalidad de bloquear el desarrollo del parásito dentro del mosquito. Para cumplir con esta meta, es indispensable identificarlas, así como entender el mecanismo por el cual el parásito es eliminado. Con base en lo anterior, investigadores en Enfermedades Transmitidas por Vectores del Centro de Investigaciones Biológicas del Noroeste, La Paz, B.C.S. y del INSP en Cuernavaca, Morelos, han realizado un estudio para identificar las proteínas que participan en la susceptibilidad o resistencia del mosquito An. albimanus durante la infección con Plasmodium.
Figura 1. Mosquito del género Anopheles spp.
Objetivos
Se tuvo como objetivo principal analizar el intestino del mosquito infectado con P. berghei, utilizando análisis proteómicos para identificar las proteínas que se sobre expresan después de una alimentación con sangre infectada con este protozoario.
Materiales y Métodos
La metodología utilizada para la identificación de las proteínas, incluyó la alimentación de los mosquitos con sangre de ratón no infectada e infectada con P. berghei, siguiendo el procedimiento descrito por Rodríguez y col. (2002). Se realizaron las disecciones de 200 mosquitos (de 3 días post-emergencia) y se separaron en dos grupos: Grupo experimental 1- 100 mosquitos alimentados con sangre de ratón sin infectar (Control). Grupo experimental 2- 100 mosquitos alimentados con sangre de ratón infectada con P. berghei. La disección se efectuó a las 24 horas post-alimentación. Posteriormente se realizó la limpieza y la cuantificación de las muestras. Por medio de la electroforesis de doble dimensión, se obtuvieron las proteínas sintetizadas durante el reto inmune. La primera dimensión o isoelectroenfoque, se llevó a cabo con la finalidad de separar las proteínas con base en su punto isoeléctrico (pI) y la segunda dimensión, mediante la electroforesis en geles de poliacrilamida SDS-PAGE al 12.5%, permitió separar las proteínas con base en su peso molecular (kDa). Los geles fueron teñidos con azul de Coomassie y escaneados. El análisis de los geles se realizó mediante el programa (Image Master 2D Platinum, Amersham Biosciences) (Serrano-Pinto et al 2010). Los spots de proteínas obtenidos en los geles, fueron cortados, digeridos con tripsina y analizados por medio de espectrometría de masas (Electrospray). La identificación de los péptidos fue reportada en términos de probabilidad con el programa Xcalibur (Thermo Fisher Scientific) en un sistema PC con Windows NT para obtener las secuencias de los péptidos sintetizados, que luego fueron comparadas contra las secuencias de proteínas homólogas en las bases de datos Swissprot y Non-redundant del NCBI (http://blast.ncbi.nlm.nih.gov/), de proteínas de An. gambiae (http://www.anobase.org/) y en el MSBLAST (http://dove.embl-heidelberg.de/Blast2/msblast.html), lo que permitió conocer el nombre y la función de cada una de las proteínas.
Resultados y Discusión
Como parte de los resultados más relevantes, se identificaron aproximadamente 200 spots de proteínas bien diferenciados. Se identificaron 21 spots de proteínas que presentaron mayor expresión en volumen en las muestras de mosquitos infectados con Plasmodium (Fig. 2).

Figura 2. Visualización de las proteínas diferenciales analizadas por electroforesis bidimensional del intestino del mosquito hembra Anopheles albimanus alimentado con: (a) sangre de ratón no infectada; (b) sangre de ratón infectada con el protozoario Plasmodium berghei. Proteínas inmunes sobre expresadas en color rojo: Spot 19- Proteína Serpina (SRPN11) ; spot 14- Proteína similar a Spaetzle (Spaetzle-like protein); spot 13- Proteína en la región, repeticiones ricas en leucina (Protein in the region, Leucine-rich repeats (LRRs) por sus siglas en inglés); spot 18- Proteína ECSIT- Señalización intermedia evolutivamente conservada en Toll (Evolutionarily Conserved Signaling Intermediate in Toll (ECSIT) por sus siglas en inglés); spot 15- Serina tipo proteasa (Serine type protease); spot 21- Serina protreasa similar a Tripsina (Trypsin-like serine protease).
Se identificaron 19 proteínas conocidas y 2 nuevas proteínas. Las diferentes categorías funcionales de las proteínas sobre expresadas en el intestino del mosquito se observan en la figura 3. Las dos clases principales de proteínas sobre expresadas, fueron la de inmunidad y defensa (34.78%) y el grupo de las proteasas digestivas (15.21%) (Serrano-Pinto et al 2010). Algunos de los péptidos identificados tuvieron similitud con las proteínas inmunes y proteasas digestivas de An. gambiae (Gorman et al 2000). Varias de estas proteínas como la proteína similar a Spaetzle un homólogo del ligando que activa el receptor Toll, la proteína Señalización intermedia evolutivamente conservada en Toll (ECSIT, por sus siglas en inglés) y una proteína adaptadora que sirve de puente para TRAF6 MEKK- 1 en la vía Toll (Kopp et al 1999) fueron inducidas en el intestino del mosquito en presencia de Plasmodium, lo que sugiere que la Vía Toll también media la respuesta antiplasmodial en este mosquito. La proteína en la región, repeticiones ricas en Leucina ((LRRs) por sus siglas en inglés), presente también en la vía Toll, tiene un papel clave en las reacciones inmunes (Osta et al 2004). La familia de los receptores Toll incluye a las moléculas transmembrana del compartimiento extracelular, donde se produce el contacto y el reconocimiento de patógenos microbianos, y el compartimento intracelular, donde se inician las cascadas de señalización que conducen a respuestas celulares (Vasselon y Detmers 2002). Se identificaron además diversas proteasas que fueron inducidas durante la infección de P. berghei (precursor de tripsina-3, quimiotripsina 1, proteasa tipo serina, y la serina proteasa similar a tripsina). La inducción del péptido Serpina (SRPN11) en las muestras infectadas en el presente estudio, sugiere que puede estar involucrado en la regulación de la respuesta inmune de An. albimanus a la infección debido a que la activación de las cascadas proteolíticas están reguladas por serpinas (inhibidores de la serina proteasa) (Danielli et al 2003, Vlachou et al 2005).
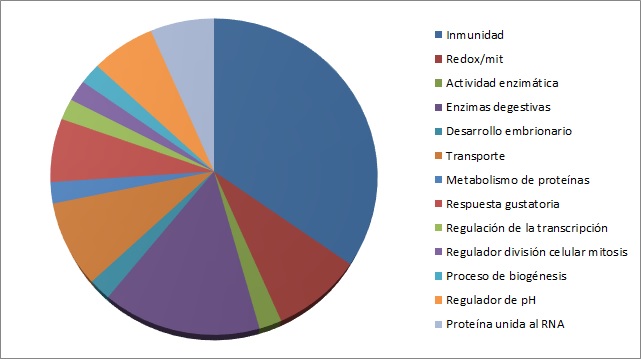
Figura 3. Diferentes categorías funcionales de las proteínas sobre expresadas en el intestino del mosquito hembra Anopheles albimanus infectado con el protozoario Plasmodium berghei.
Conclusiones
En conclusión, con base en el análisis de estas proteínas y de sus funciones, se aporta nuevo conocimiento sobre mecanismos moleculares del desarrollo de P. berghei en An. albimanus que podrán ser utilizadas para establecer moléculas blanco que permitan el bloqueo del desarrollo del parásito en su vector mediante el silenciamiento de los genes que sintetizan a estas proteínas al inyectar RNA de interferencia (RNAi) en estos organismos, lo que puede servir como base para el desarrollo de mosquitos trangénicos resistentes a la transmisión.
Impacto Socioeconómico
Impacto socioeconómico
En todo el país se trabaja en establecer campañas efectivas de erradicación y manejo de este vector, orientadas a minimizar en lo posible el impacto negativo que se genera en el humano por esta enfermedad, sin descartar los altos costos anuales asignados para su control, por lo que este estudio servirá para establecer las bases para el desarrollo de nuevas estrategias de manejo y control que llevarán al objetivo final de erradicación de esta enfermedad en México y en otros países donde An. albimanus es el vector.
Referencias
Boletín de Epidemiología, SSA, México, 2011.
Collins, F.H., 1994. Prospects for malaria control through the genetic manipulation of its vector. Parasitol. Today 10, 370–376.
Danielli, A., Kafatos, F.C., Loukeris, T.G., 2003. Cloning and characterization of four Anopheles gambiae serpin isoforms, differentially induced in the midgut by Plasmodium berghei invasion. J. Biol. Chem. 278, 4184e4193.
Dimopoulos G., Seeley D., Wolf A. and Kafatos F.C. 1998. Malaria infection of the mosquito Anopheles gambiae activates immune-responsive genes during critical transition stages of the parasite life cycle. EMBO J, 17:6115-6123
Gorman, M.J., Andreeva, O.V., Paskewitz, S.M. 2000. Molecular characterization of five serine protease genes cloned from Anopheles gambiae hemolynph. Insect Biochem. Mol. Biol. 30, 35-46.
Ito, J, Ghosh, A, Moreira, L, Wimmer, E. A., Jacobs-Lorena, M. 2002. Transgenic anopheline mosquitoes impaired in transmission of a malaria parasite. Nature. 417: 452-455.
Kaslow, D.C. 2002. Transmission-blocking vaccines. Chem. Immunol. 80:287-307.
Kopp, E., Medzhitov, R., Carothers, J., Xiao, Ch., Douglas, I., Janeway, C.A. Ghosh, S. 1999. ECSIT is an evolutionarily conserved intermediate in the Toll/IL-1 signal transduction pathway. Genes & Develop. 13, 2059–2071.
Mendis K, Sina BJ, Marchesini P, Carter R. 2001. The neglected burden of Plasmodium vivax malaria. Am. J. Trop. Med. Hyg. 64: 97-106.
OMS. 2012. Organización Mundial de la Salud. Estadísticas Sanitarias Mundiales 2012.
Osta, M.A., Christophides, G.K., Vlachou, D., Kafatos, F.C. 2004. Innate immunity in the malaria vector Anopheles gambiae: comparative and functional genomics. J. Exper. Biol. 207, 2551-2563.
Rodrigues, J., Agrawal, N., Sharma, A., Malhotra, P., Adak, T., Chauhan, V.S., Bhatnagar, R.K., 2007. Transcriptional analysis of an immune-responsive serine protease from Indian malarial vector, Anopheles culicifacies. BMC Mol. Biol. 15, 8e33.
Rodríguez, M.C., Margos, G., Compton, H., Ku, M., Lanz, H., Rodríguez, M.H., Sinden, R., 2002. Plasmodium berghei: routine production of pure gametocytes extracellular, gametes, zygotes and ookinetes. Exp. Parasitol. 101, 73e76.
Rodríguez-López MH, Loyola-Elizondo EG, Betanzos-Reyes AF, Villarreal-Treviño C, Bown DN. 1994. Control focal del paludismo. Gaceta Médica de México, 130: 313-319.
Serrano-Pinto, V., Acosta M., Luciano-Bazán, D., Hurtado-Sil, G., Ferreira-Batista, C., Martínez-Barnetche, J., Lánz-Mendoza, H. 2010. Differential expression of proteins in the midgut of Anopheles albimanus infected with Plasmodium berghei. Insect Biochem. Mol. Biol. 40(10): 752-758. Doi: 10.1016/j.ibmb.2010.07.011.
Uribarren Berrueta, T. 2014. Paludismo o Malaria. Departamento de Microbiología y Parasitología, Facultad de Medicina, UNAM.
Vasselon, T and Detmers, P.A. 2002. Toll Receptors: a Central Element in Innate Immune Responses. Infect Immun. 70(3): 1033–1041.
Vlachou, D., Schlegelmilch, T., Christophides, G.K., Kafatos, F.C., 2005. Functional Genomic analysis of midgut epithelial responses in Anopheles during Plasmodium invasion. Current Biol. 15, 1185e1195.
Xu,W.Y., Huang, F.S., Hao, H.X., Duan, J.H., Qiu, Z.W., 2006. Two serine protease from Anopheles dirus heamocytes exhibit changes in transcript abundance after infection of an incompatible rodent malaria parasite, Plasmodium yoelii. Vet. Parasit. 139, 93e101.
